Detección de Melanoma con Deep Learning: De la Imagen al Diagnóstico Asistido
En esta entrada explico cómo he desarrollado un modelo de clasificación de lunares benignos y malignos a partir de imágenes, y cómo estoy evolucionando el proyecto hacia un enfoque más clínico usando criterios ABCDE del melanoma.
PROYECTOS
6/17/20252 min read
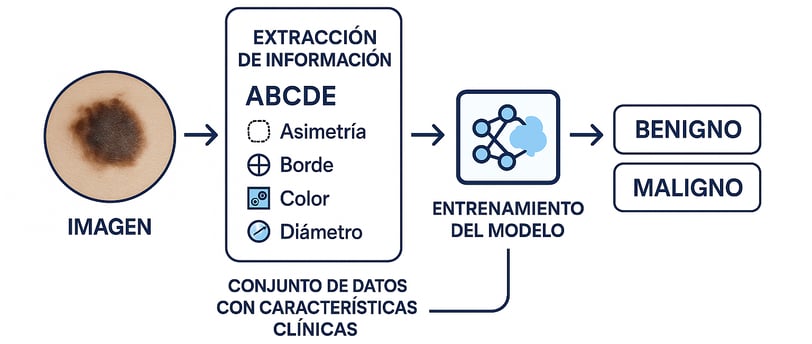
Clasificación de lunares con Deep Learning: de CNNs simples a criterios clínicos ABCDE
Uno de mis primeros proyectos personales en visión por computadora ha sido aplicar redes neuronales convolucionales (CNNs) para clasificar imágenes de lunares benignos y malignos. Es un tema sensible e interesante desde el punto de vista biomédico, así que decidí ir más allá de una simple red funcional.
🧪 Primera versión del proyecto
En la versión inicial:
Usé ImageDataGenerator con augmentación para compensar la escasez de imágenes.
Entrené una CNN desde cero en TensorFlow/Keras.
Utilicé carpetas separadas para imágenes benignas y malignas.
Apliqué callbacks como EarlyStopping y ModelCheckpoint.
Obtuve resultados funcionales, pero la precisión del modelo no fue lo suficientemente buena para un caso clínico real, y empecé a notar los límites de usar solo una CNN "black box" con augmentaciones agresivas.
🔄 Redefiniendo el enfoque: de CNN a un modelo clínicamente orientado
Decidí replantear el pipeline con una aproximación más rigurosa y alineada con el análisis médico:
1. Evitar modificaciones que deformen las imágenes
Las alteraciones por rotaciones, zoom o desplazamientos pueden comprometer estructuras clave en lesiones cutáneas. Prefiero trabajar con las imágenes originales.
2. Crear un archivo CSV con metadatos
Estoy construyendo un CSV que contenga, para cada imagen:
Nombre del archivo
Etiqueta (benigno/maligno)
Y ahora también características clínicas extraídas automáticamente
3. Extraer métricas basadas en el criterio ABCDE del melanoma:
Asimetría: diferencia de forma entre mitades.
Bordes: grado de irregularidad (usando contornos y medidas geométricas).
Color: cantidad de tonos dominantes y variabilidad en el histograma.
Diámetro: medida en píxeles.
(En futuro) Evolución: si se dispone de imágenes temporales.
Estas características serán computadas con funciones específicas en Python y OpenCV, y almacenadas en el CSV para su análisis posterior.
4. Entrenar un nuevo modelo con datos estructurados
Con el CSV, utilizaré algoritmos como:
Random Forests o XGBoost (alta precisión y explicabilidad),
O incluso una arquitectura híbrida CNN + capas densas tabulares.
🔍 Próximos pasos
Finalizar el extractor automático de características ABCDE.
Evaluar el rendimiento de diferentes modelos sobre los datos estructurados.
Documentar todo el proceso en GitHub, con notebooks y comparativas.
📁 Repositorio del proyecto
Todo el código está disponible (y en evolución constante) en mi GitHub:
👉 [enlace al repo]
🔚 Reflexión final
Este proyecto ha sido una gran forma de profundizar en visión por computador, clasificación binaria, buenas prácticas de entrenamiento y, sobre todo, en cómo adaptar los modelos a los requisitos clínicos reales. No se trata solo de que funcione: se trata de que tenga sentido y se pueda confiar en él.
Santiago Fernández
🧠 Diseño que resuelve cosas que importan
🖨️ 3D · 📐 CAD · 🛠️ Prototipado técnico
Ingeniería
Freelance
© 2025. All rights reserved.
